整理下一些评价指标,一般数据是从下面这种frequency表开始统计的

Sensitivity
灵敏度(Sensitivity)是指在所有实际患病的人中检测呈阳性的比例;其可认为对微小的变化有多敏感,比如:
胰腺炎具有淀粉酶这个特征,如果未检测到该特征,则该疾病可能并不存在并且可以被排除。这是因为:淀粉酶的检测是高度敏感的,因为能够在血液中检测到非常少量的淀粉酶。所以,存在“低于检测阈值”的淀粉酶的几率是很小。因此,一个阴性的结果意味着:1. 淀粉酶是存在的,但是数量非常少,以至于测试无法检测到它(不太可能,因为这个能检测到微小的变化)。2. 淀粉酶根本不存在(更有可能)。
公式:Sensitivity = True positive / (True positive + false negative),即 a / (a + c)
Specificity
特异度(Specificity)是指在所有实际没有该疾病的人中检测呈阴性的比例,其有助于在疾病呈阳性时排除疾病,比如:
尿路感染患者尿液中的亚硝酸胺在其他疾病中极少出现,因此这个检测指标对尿路感染具有很高的特异度;但是,其检测阳性并不说明肯定有尿路感染,因为高特异度的检测不考虑疾病的患病率
公式:specificity = True negative / (True nagetive + false positive),即 d / (b + d)
从上可看出,Sensitivity和Specificity是检测的特性,其于患病率没什么联系;但对于positive predictive value (PPV) and negative predictive value (NPV)而言,其则是跟患病率相关,因此可以从临床的角度来说 患者患某种疾病的可能性有多大
Positive predictive value (PPV)
阳性预测值(PPV)是指检测阳性后,个体真正患有特定疾病的可能性
公式:PPV = True positive / (True positive + false positive),即 a / (a + b)
Negative predictive value (NPV)
阴性预测值(NPV)是指检测阴性后,个体确实不会患有特定疾病的可能性
公式:NPV = True nagative / (True negative + false negative),即 d / (c + d)
Summary
- 假定一个检测灵敏度和特异度保持不变,当患病率降低时,PPV则会随之降低,这是因为对于发现每一个真阳性会有更多的假阳性,这就相当于大海捞针;而NPV则会随之升高,这是因为对于发现每一个假阴性会有更多的真阴性,患病率降低了,假阴性也随之减少了
- 因此如果我们在高危人群中进行诊断试验,我们能够得到更为高效的结果,避免人力、物力的浪费。同时,诊断试验在临床应用中,我们也需要根据患病率来深入分析结果;还可以们在临床上拿到诊断试验结果时,应根据其人群患病率,综合判断结果的真实性。
- 在相同情况下,提高特异度比提高灵敏度更有助于提高阳性预测值;这是因为当患病率比较低时(一般情况下,人群患病率不会太高),整个人群中患者数很少,即使提高灵敏度也不会大幅增加真阳性人数,所以对阳性预测值的改变较小。同时,非患者数很多,提高特异度会大幅减少假阳性人数,所以对阳性预测值的改变较大。
- 如果一项诊断试验的灵敏度比较低,则会有较多的假阴性,延误患者的就诊,进而影响病程的发展和愈后;如果特异度较低,则会有较多的假阳性,进而浪费医疗资源和引起患者的恐慌焦虑
参考自:https://zhuanlan.zhihu.com/p/25164465
Likelihood Ratio(PLR/NLR)
首先是likelihood ratio的定义:

具体的讲:
阳性似然比 = 实际患病的人中检测呈阳性的比例 / 实际没有患病的人中检测呈阳性的比例,则公式:PLR = sensitivity / (1 – specificity)
阴性似然比 = 实际患病的人中检测呈阴性的比例 / 实际没有患病的人中检测呈阴性的比例,则公式:NLR = (1 – sensitivity) / specificity
似然比(LR)是可以同时反应敏感度和特异度的复合指标;假如PLR等于10,说明当诊断试验结果为阳性时,判定患病的可能性是不患病可能性的10倍;若NLP为0.01,意味着当诊断试验结果为阴性时,判定患病的可能性仅为不患病的1/100
- LR=[0-1]时,发病证据减少,接近于0则会降低患病的可能性
- LR=1时,无诊断价值
- LR>1时,发病证据增加,比1越大则患病的可能性就越大
LR还与Post-Test Odds以及Pre-Test Odds有一定关系:Post-Test Odds = Pre-Test Odds * LR,比如:
按照实践经验,70%因发烧和关节痛从里约热内卢返回的患者会患有寨卡病;假如某个患者验血寨卡病为阳性,似然比为6,那么这个患者患有寨卡病毒的可能性有多大
- Step 1: Convert the pre-test probability to odds,
0.7 / (1 – 0.7) = 2.33 - Step 2: Use the formula to convert pre-test to post-test odds,
Post-Test Odds = Pre-test Odds * LR = 2.33 * 6 = 13.98 - Step 3: Convert the odds in Step 2 back to probability,
(13.98) / (1 + 13.98) = 0.93
以上LR参考:https://www.statisticshowto.com/likelihood-ratio/
Area Under Curve(AUC)
AUC是指受试者工作特征曲线下面积,一般可通过计算ROC曲线下的面积得出AUC值
ROC 曲线(接收者操作特征曲线)是一种显示分类模型在所有分类阈值下的效果的图表,其主要涉及两个参数:真阳性率(TPR)和假阳性率(FPR);ROC 曲线用于绘制采用不同分类阈值时的 TPR 与 FPR。降低分类阈值会导致将更多样本归为正类别,从而增加假正例和真正例的个数;因此曲线越靠左上角,灵敏度和特异度就越好,AUC值也会相应的变大,即指标的诊断能力就越好
AUC取值在0.5-1之间;一般认为,AUC在0.50-0.70为较低准确度,在0.71-0.90之间为中等准确度,高于0.90则为高准确度
Harrell' Concordance Index(C-index)
C-index主要用于计算生存分析中的COX模型预测值与真实之间的区分度(discrimination)
C-index的计算方法是: 把所研究的资料中的所有研究对象随机地两两组成对子。以生存分析为例,对于一对病人,如果生存时间较长的一位,其预测生存时间长于生存时间较短的一位,或预测的生存概率高的一位的生存时间长于生存概率低的另一位,则称之为预测结果与实际结果一致:
1. 产生所有的病例配对。若有n个观察个体,则所有的对子数应为Cn2(组合数) 2. 排除下面两种对子:对子中具有较小观察时间的个体没有达到观察终点及对子中两个个体都没达到观察终点;剩余的为有用对子 3. 计算有用对子中,预测结果和实际相一致的对子数, 即具有较坏预测结果个体的实际观察时间较短 4. 计算 C-index = 一致对子数/有用对子数
一般我习惯用R的survival包来计算Cox回归模型以及相应的C-index值
Net Reclassification Improvement(NRI)
AUC值只能说明指标的诊断能力的大小,但我们一般关心的是指标某个界值下诊断正确与否;例如指标A和指标B对应的AUC值分别为0.76和0.77,两者的AUC值并没有统计差异(两个模型的AUC比较采用Z检验),但如果选择某个界值,B指标诊断准确度高于A指标,这时就需要NRI来判断
NRI(Net Reclassification Improvement,净重分类改善指数)用于比较两个指标在某个界值下的诊断能力,一个指标比另外一个指标诊断准确率是否提高,计算公式:NRI= (Test2_sensitivity + Test2_specificity) - (Test1_sensitivity + Test1_specificity),此公式代表指标2比指标1分类改善的比例,若所得到的NRI大于0则为正改善(预测能力有所提高),小于0则为负改善(预测能力下降),等于0则为无改善
可用nricens包的nribin( )函数计算,并根据不同界值求得NRI的点估计值和95%置信区间
Integrated Discrimination Improvement(IDI)
NRI有个不足在于只考虑了在某个界值下的诊断能力改善情况,不能考察模型的整体改善情况,因此我们需要另外一种指标:IDI(Integrated Discrimination Improvement,综合判别改善指数),由于其考虑了不同界值下的情况,可以用来反映模型的整体改善状况
虽然AUC也考虑到了不同界值,但是AUC的改善情况在临床中不易解释,IDI也因此弥补了AUC的缺陷,可以形象地展示研究对象被准确重新判别的比例
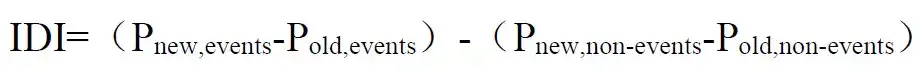
其中Pnew,events、Pold,events表示在患者组中,新模型和旧模型对于每个个体预测疾病发生概率的平均值,两者相减表示预测概率提高的变化量,对于患者来说,预测患病的概率越高,模型越准确,因此差值越大则提示新模型越好。
而Pnew,non-events、Pold,non-events表示在非患者组中,新模型和旧模型对于每个个体预测疾病发生概率的平均值,两者相减表示预测概率减少的量,对于非患者来说,预测患病的概率越低,模型越准确,因此差值越小则提示新模型越好。
最后,将两部分相减即可得到IDI,总体来说IDI越大,则提示新模型预测能力越好。与NRI类似,若IDI>0,则为正改善,说明新模型比旧模型的预测能力有所改善,若IDI<0,则为负改善,新模型预测能力下降,若IDI=0,则认为新模型没有改善。
一般会用R的nricens包中nribin()函数,增加bootstrap参数可得到置信区间
以上均整理自网上资料
参考资料
https://kuaibao.qq.com/s/20180516G0F8XX00?refer=spider
https://www.msdmanuals.com/medical-calculators/LikelihoodRPData-zh.htm https://geekymedics.com/sensitivity-specificity-ppv-and-npv/ https://www.mediecogroup.com/method_topic_article_detail/140/ https://zhuanlan.zhihu.com/p/25164465
本文出自于http://www.bioinfo-scrounger.com转载请注明出处