ROC和AUC平时用的比较多,但是其真正的原理和计算过程却了解的不多,因此做个整理
照抄自网上的一个例子,加深下理解:
比方说在一个10000个人的数据集中,有100个人得了某种病症,你的任务是来预测哪些人得了这种病症。你预测出了200人得了癌症,其中: * TN,True Negative:没有得癌症并且你也预测对没有得癌症的有9760人 * TP,True Positive:得了癌症而且你也预测出来的病人有60人 * FN,False Negative:得了癌症但是你没有预测出来的病人有40人 * FP,False Positive:没有的癌症但是你预测得了癌症的有140人
那么:
- True Positive Rate(TPR): 60/(60+40)=0.6
- False Positive Rate(FPR): 140/(9760+140)=0.0141
- accuracy: (9760+60)/10000=0.982
- precision: 60/(60+140)=0.3
- recall: 60/100=0.6 从上述例子可看出,几个重要指标的含义如下(盗图):

简单的说,在上述癌症检测中(正反例极度不平衡的情况下),accuracy意义不大,从结果中可看出,因为正例比较少,所以检测再不怎么准确,其accuracy值都很高。recall则是相对于真实情况而言的,你正确检测了60例癌症病人,总癌症病人有100例,那么recall则为60%。precision则是相对于模型预测情况而言的,你预测了200例癌症病人,其中预测准确的有60例,那么precision则为30%;所以我们是希望能看到模型的recall和precision都很高,但是一般两者难以同时达到最优值,需要做一个权衡
有时我们还会见到sensitivity和specificity两个概念:
- sensitivity = recall = True Positive Rate
- specificity = 1- False Positive Rate
也就是说想要sensitivity高一点相当于要True Positive Rate高一点,要specificity高一点相当于False Positive Rate低一点
话说为了权衡recall和precision,对于评判二分类器的优劣,可以使用ROC(Receiver Operating Characteristic)曲线以及AUC(Area Under roc Curve)指标;ROC平时用的很多,其计算方式整理如下:
先清楚ROC曲线的几个概念,横坐标是FPR,纵坐标是TPR,而图中每个点则是不同"截断点"下计算的FPR和TPR,截断点相当于模型对于样本的概率输出,也可以说是打分score

以一个简单的模拟数据来计算下ROC曲线每个点的值
- 先有一组真实分类,比如0,1,0,1;然后一组模型预测的打分(概率),比如:0.2,0.3,0.5,0.8
- 依据上述打分依次计算每个样本输出概率下的FPR和TPR
- 首先截断点为0.2,当概率大于等于0.2时,则预测为正例,因此4个样本均为正例(1),这时的FPR=1,TPR=1
- 接着截断点为0.3,因此当概率大于等于0.3时预测为正例,因此样本1预测为反例,样本2-4为正例,这时的FPR=0.5,TPR=1
- 以上述方法计算截断点0.5和0.8,当为0.5时,FPR=0.5,TPR=0.5;当为0.8时,FPR=0,TPR=0.5
从上述计算方法可看出,ROC一般是只用于二分类器的预测评价;我们也可以用工具来检验下上述的计算结果,Python可以用sklearn,R可以用ROCR包或者pROC包,这里以ROCR包来检验下上述计算结果:
library(ROCR)
y <- c(0,1,0,1)
p <- c(0.2,0.3,0.5,0.8)
pred <- prediction(p, y)
perf <- performance(pred, "tpr", "fpr")
perf
> perf
An object of class "performance"
Slot "x.name":
[1] "False positive rate"
Slot "y.name":
[1] "True positive rate"
Slot "alpha.name":
[1] "Cutoff"
Slot "x.values":
[[1]]
[1] 0.0 0.0 0.5 0.5 1.0
Slot "y.values":
[[1]]
[1] 0.0 0.5 0.5 1.0 1.0
Slot "alpha.values":
[[1]]
[1] Inf 0.8 0.5 0.3 0.2x.values对应FPR,y.values对应TPR, alpha.values对应预测打分cutoff,结果跟上面完全一致,然后简单做个ROC图,个人喜欢pROC包的图。。。
library(pROC)
modelroc <- roc(y,p)
plot(modelroc, print.auc=TRUE, auc.polygon=TRUE)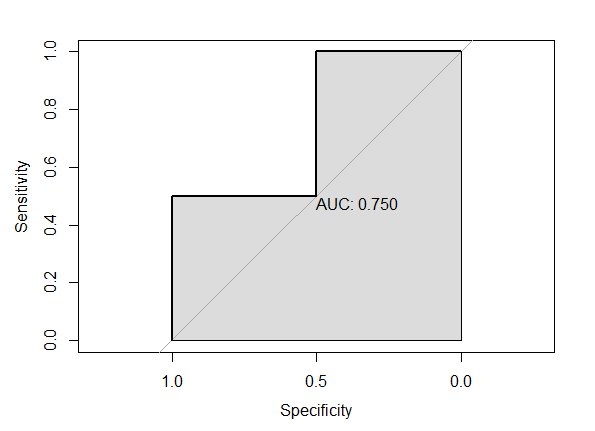
上述的图中的AUC值就相当于ROC曲线的所覆盖的面积,可以从ROC曲线看出AUC值越大,其分类效果越好;如果AUC等于0.5,则相当于做个预测结果跟随机的没啥差别
至于怎么计算AUC:
- 从早期的计算ROC曲线下的面积
- 现在的可以通过计算正例score大于反例score的概率;比如上面例子中正例有2个(M),反例有2个(N),那么总共有2*2=4对([0.3,0.2],[0.8,0.2],[0.3,0.5],[0.8,0.5])正反样本对,同时通过其模型预测的score可看出正例score大于反例的有3对([0.3,0.2],[0.8,0.2],[0.8,0.5]),因此AUC则为3/4=0.75
- 当样本比较多时,上述算法的复杂度过高
O(N*M),有人提出一个简单的算法:对score从到小排序,最大的score排序索引为n,最小的则为1;然后将正例的索引求和,减去正例-正例这种组合的个数M*(M+1)/2;最后除以M*N。以上述为例:正例的排序索引为2和4,M和N都为2,因此AUC=(2+4-2*(2+1)/2)/(2*2)=0.75
从上述可看出,只要理解了ROC和AUC的原理,其实计算方法都蛮简单的
参考资料:
AUC的计算方法及相关总结
ROC与AUC的定义与使用详解
本文出自于http://www.bioinfo-scrounger.com转载请注明出处