最近看了一些Galaxy的一些官方文档,学习下如何在Galaxy这个开源生信云平台上构建一些分析工具,最后不是太令人满意;整体上Galaxy还是很好的一个平台工具,其开源平台再加上开发者们提供的开源分析流程能很好的搭建一些生信分析流程,其也设定好了一个前段-后端的框架,用户可以通过注册并登陆,以及保存数据集以及分析的输入/输出文件,也可以通过html文件设定登陆界面等等 主要的一些Galaxy操作说明都在https://galaxyproject.org/admin/中
比如用Tool Shed安装工具,可以看文档Installing Tools into Galaxy,比如常见的bwa、hisat2等等
但通常一般会本地化Galaxy,那么需要将自己的登陆账户设置为管理员,这样才能通过Tool Shed安装软件,如修改配置文件galaxy.yml,将其中的admin_users后面加上自己的注册邮箱即可,然后重启Galaxy,然后点击管理员后(我是中文界面。。。),界面如下:
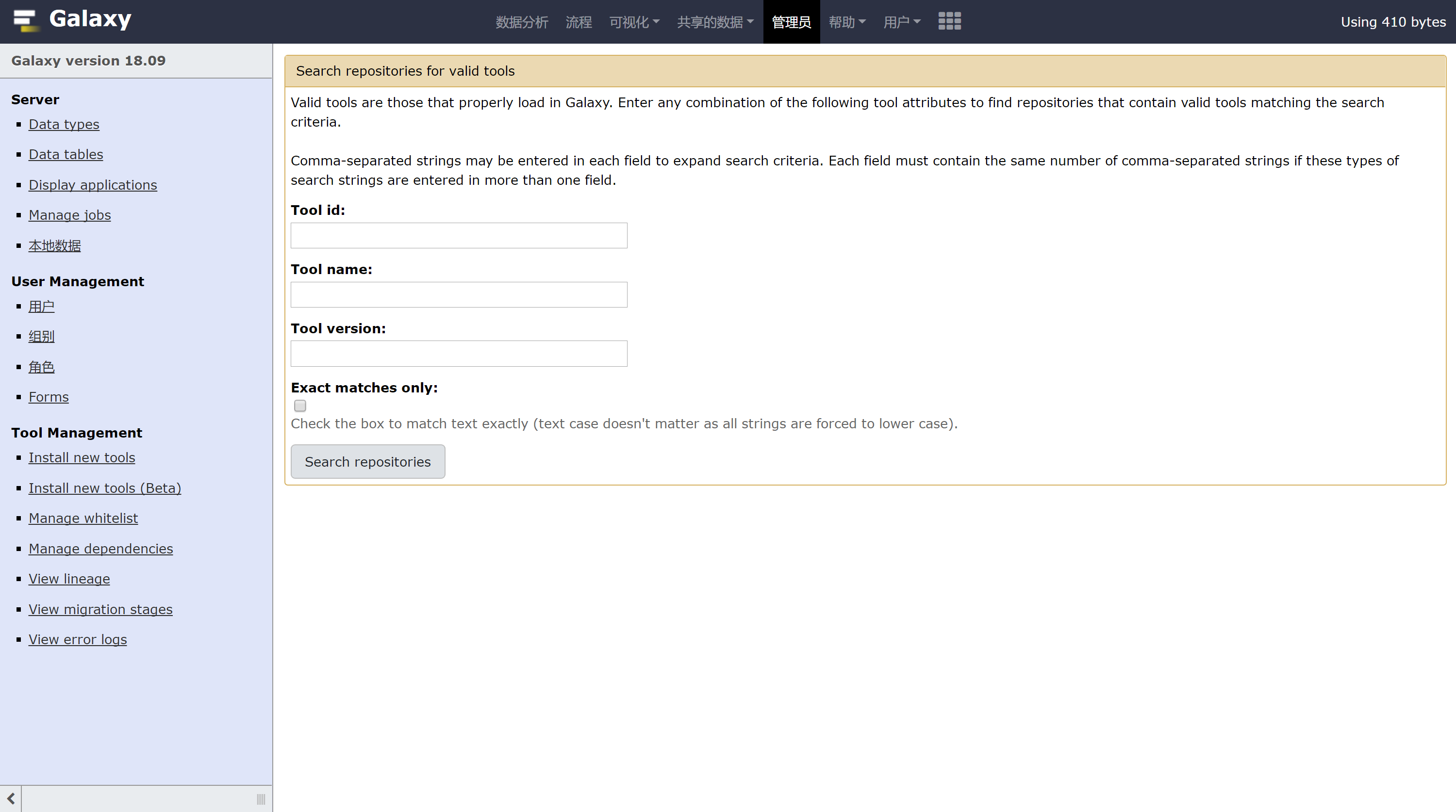
如果从shed tools上安装的软件需要一些依赖软件或者想直接使用本地上已经安装的版本,那么可以参考Tool Dependencies,其实就是将软件执行文件的路径放到系统环境变量中
对于Tool Panel的管理可以参照Tool Panel Administration,明白其布局规则(config/tool_conf.xml),大致格式如下():
<?xml version='1.0' encoding='utf-8'?>
<toolbox monitor="true">
<section name="MyTools" id="mTools">
<tool file="myTools/toolExample.xml" />
</section>
<section id="getext" name="Get Data">
<tool file="data_source/upload.xml" />
</section>
</toolbox>界面如下:

如果想自定义本地软件或者一些脚本到Galaxy中的话,这个是我最为关注的,可以参考Adding custom tools to Galaxy
一般步骤如下,以计算fasta文件的GC含量为例:
- 在tools文件夹下新建myTools目录
- 写一个计算GC含量的perl脚本,如:
toolExample.pl(也可以是Python脚本或者R脚本等等,但必须是可以用命令行支持的,其他自定义软件也是类似操作原理) - 在
toolExample.xml配置脚本/软件执行方式以及输入/输出变量 - 将上述两个文件放到myTools目录下,重启Galaxy
toolExample.pl和toolExample.xml示例参照上述链接下的文章,有很详细的解释说明
Galaxy还支持自定义绘图工具,如Creating a histogram tool tutorial,具体原理跟上面那个自定义工具一样,但是其支持的绘图工具有点非主流。。。
我查看了Galaxy内置的bar_chart绘图工具脚本,其是用Python调用Gnuplot来实现绘图的,我最开始想用R来实现图形展示,但是无法输出图片到Galaxy上,晚上搜索了好久也没查到Galaxy和R绘图的关联资料
那么Galaxy是如何来实现可视化的呢,其是利用javascript来实现可视化的,其示例都在Galaxy平台上的可视化模块内,可参照Visualization with Charts,有兴趣的可以试试。。。
除了上述安装Galaxy工具外,其还支持用Planemo来build tools,如参考Building Galaxy Tools Using Planemo
整体上Galaxy还是一个很好用的开源生信平台工具,但是个人觉得由于其在某些方面显的很方便,从而使得其看起来比较的臃肿以及个性化展示方面显得比较的约束,这点比起Shiny可能做的更加的好些,毕竟Shiny更加贴近网页工具
Galaxy学习资料网上比较少,主要还是得依靠官方文档,推荐一些中文版资料:
Galaxy生物信息平台:charts 自定义可视化
利用Docker搭建Galaxy
本来还想深入了解下Galaxy,看看是否能做个简易版的生信云平台搞搞,但是了解了后,觉得可能不太合适,变成了从入门到放弃系列。。。
后续看看学下Python的web后端框架,从传统思路上来试试,比如简单的搭建个登录界面以及数据库管理,然后再搭配上一些前端网页以及一些shiny可视化?
本文出自于http://www.bioinfo-scrounger.com转载请注明出处