现在CNV分析方法有很多,但是随着NGS成本的降低,高深度的测序下背景下,read count(depth)方法逐渐变为各个分析软件的主流。最近在看GATK的CNV分析方法,其中在创建PoN(PanelofNormals)和denoise过程都使用了PCA(Principal Component Analysis)的奇异值分解(Singular Value Decomposition, SVD)方法,因此有比较对PCA思路做个梳理以及了解下常见的如何使用PCA来降噪(denoise) R语言中,PCA一般常用的基础函数是prcomp和princomp,两者有些差别:
- prcomp适用于R mode和Q mode,而princomp只适用于R mode
- prcomp基于奇异值分解(SVD),求最小二乘的优化解(最小化的结果要求寻找一个正交矩阵AB,记作H[q],H[q]为投影矩阵,把每个点X[i]投影到q维结构H[q]x[i],这是由V[q]的列限定的x[i]的正交投影)
- princomp是基于相关系数矩阵的特征值分解,方差最大化方法(本质上是求原始变量协方差/相关系数矩阵的特征值和特征向量,第i主成分对应着协方差的第i大特征值,投影方向为该特征值对应的特征向量,而特征值等于相应主成分的方差,因此代表着对应主成分分解原始信息的多少)
- prcomp在参数中如果设定了
scale=T,那么如果princomp结果想与prcomp保持一致,则princomp函数需加上cor=T参数(其实原因在于cor和cov之间刚好相差了一个scale)
R mode是指基于variable分析,一般来研究variable之间的关系,当数据集的observation数目大于variable时;Q mode是指基于observation分析,一般来研究observation之间的的关系,当数据集的varibale数目大于observation时
从R代码的角度来实现下PCA实现思路(原理部分可以看图文并茂的PCA教程),并与prcomp函数的结果比较下,代码参考PRINCIPAL COMPONENTS ANALYSIS (PCA);使用自带数据集USArrests作为测试对象
先是prcomp函数通过奇异值分解来实现PCA分析,res1$sdev是特征值的平方根(由矩阵的奇异值计算得出的),res1$rotation是变量的loading矩阵(每列是对应主成分的特征向量),res1$x是PCA projections ("scores")
data(USArrests)
dim(USArrests)
res1 <- prcomp(USArrests, scale = T)
sd <- res1$sdev
loadings <- res1$rotation
scores <- res1$x然后是R代码实现
R <- cor(USArrests)
# find the eigenvalues and eigenvectors of correlation matrix
myEig <- eigen(R)
# eigenvalues stored in myEig$values
# eigenvectors (loadings) stored in myEig$vectors
loadingsLONG <- myEig$vectors
rownames(loadingsLONG) <- colnames(USArrests)
# calculating singular values from eigenvalues
sdLONG <- sqrt(myEig$values)
# transforming data to zero mean and unit variance
X <- apply(USArrests, 2, function(x){
(x - mean(x))/sd(x)
})
# calculating scores from eigenanalysis results
scoresLONG <- X %*% loadingsLONG比较两者的前后两种方法的差异(其实是很小的)
range(sd - sdLONG)
range(loadings - loadingsLONG)
range(scores - scoresLONG)如果想降噪,则简单的说相当于要做了以下步骤:
- 从n维降到k维
- reconstruct初始变量,相当于将降维后的数据升到n维,即:PCA reconstruction=PC scores*EigenvectorsT+Mean >什么是重建(reconstruct), 那么就是找个新的基坐标, 然后减少一维或者多维自由度。 然后重建整个数据。 好比你找到一个新的视角去看这个问题, 但是希望自由度小一维或者几维。主成分分析PCA算法:为什么去均值以后的高维矩阵乘以其协方差矩阵的特征向量矩阵就是“投影”?
- 得到新的数据集为去噪后的数据集
下面用一个示例来说明下使用不同k维下的区别,用R代码来说明下如何进行降噪。参考一篇文章R Basics: PCA with R和帖子How to reverse PCA and reconstruct original variables from several principal components?以及How to perform dimensionality reduction with PCA in R
测试数据是Kaggle上的一个测试数据集MNIST
require(data.table)
mnist_data=fread('../Desktop/all/train.csv')然后对数据进行预处理,去除掉对应的观测值是常量的变量(具体原因可看上述那篇文章)
mnist_data2 <- mnist_data[,-(which((1:784)%%28<=2|(1:784)%%28>=26|(1:784)%/%28<=2|(1:784)%/%28>=26)+1), with=F]使用prcomp函数进行PCA分析,并从解释方差比例角度来看下不同主成分的重要性
pca_res <- prcomp(mnist_data2[,(2:ncol(mnist_data2)), with=F], center = T, scale. = F)
plot(cumsum(pca_res$sdev^2)/sum(pca_res$sdev^2)*100,
ylab = 'Cumulative proportion of variance explained', xlab = "Principal Component")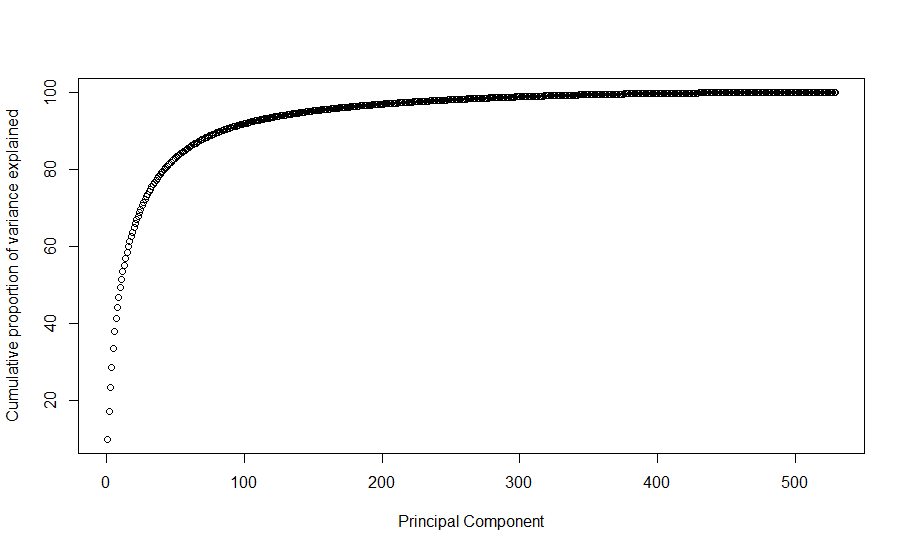
最后看下新投影(或者说reconstruct后的)数据集
pc_use <- 300
trunc <- pca_res$x[,1:pc_use] %*% t(pca_res$rotation[,1:pc_use])
trunc <- scale(trunc, center = -pca_res$center, scale = pca_res$scale)
dim(trunc)
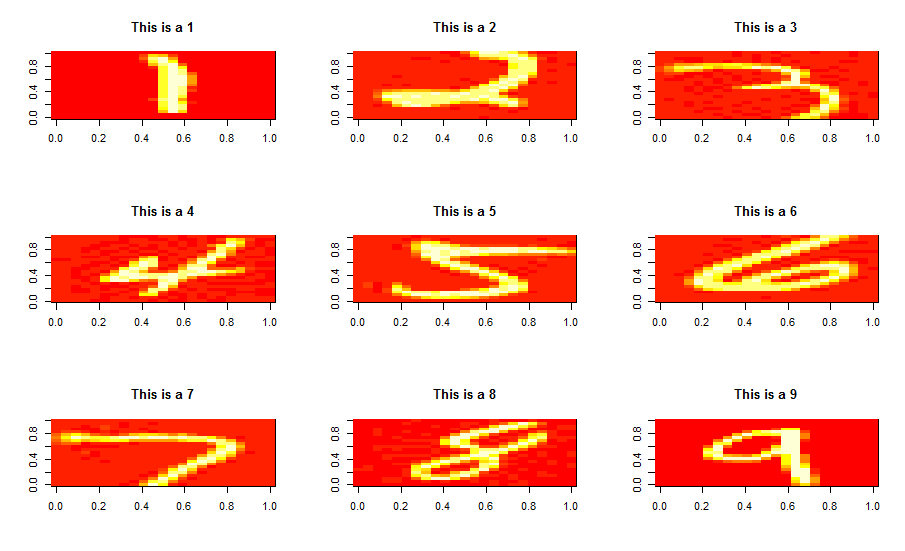
res <- data.table(mnist_data2$label, trunc)并与初始数据集的图片做个比较看下,原数据集如下:
par(mfrow=c(3,3))
for (i in 1:9) {
##Changing i-th row to matrix
set.seed(123456)
rad <- sample(which(mnist_data[,1] == i), 1)
mat <- matrix(as.numeric(mnist_data[rad, 785:2]), nrow = 28, ncol = 28, byrow = F)
##Inverting row order
mat <- mat[nrow(mat):1,]
##plot
image(mat,main=paste0('This is a ', i))
}
再看下只取前300个主成分的新投影数据集(其去除了不太重要的一些维度,降低了维度)
par(mfrow=c(3,3))
for (i in 1:9) {
set.seed(123456)
rad <- sample(which(res[,1] == i), 1)
mat <- matrix(as.numeric(res[rad, 530:2]), nrow = 23, ncol = 23, byrow = F)
mat <- mat[nrow(mat):1,]
image(mat,main=paste0('This is a ', i))
}
从图像降噪的角度来看,上述的例子不太好,可以看这篇用Python来实现PCA降噪的文章机器学习:PCA(降噪),来实现对高斯噪声的去除
总之,使用PCA等手段可以有效的去除一些噪声,压缩等
其他参考文章:
Reversing PCA back to the original variables [duplicate]
http://statmath.wu.ac.at/~hornik/QFS1/principal_component-vignette.pdf
一篇深入剖析PCA的好文
R语言主成分分析——prcomp VS princomp
R语言主成分分析——prcomp VS princomp
初识R语言——PCA的实现
本文出自于http://www.bioinfo-scrounger.com转载请注明出处