在学习全基因组call variant时,用过snpEFF/Annovar两款对vcf文件注释的软件,如两篇博文所记录的简单学习snpEFF注释vcf-笔记和Annovar注释vcf-笔记,那时并未对结果进一步的解读,比如:Sequence Ontology这个概念
Annovar
Annovar的Gene-based annotation的结果有两个文件: >1. 在外显子位点上的突变注释:XX.exonic_variant_function
>2. 所有位点上的突变注释:XX.variant_function
XX.variant_function文件一般关心前两列,后面几列均是变异位点的一些信息(因为我那时没有选择其他snp相关数据库,只是Gene-based annotation的话,所得信息会比较少): 
第一列是变异所在基因组的位置,如:exonic、splicing、ncRNA等;其优先级:exonic = splicing > ncRNA> > UTR5/UTR3 > intron > upstream/downstream > intergenic
如下图官方文档所示:
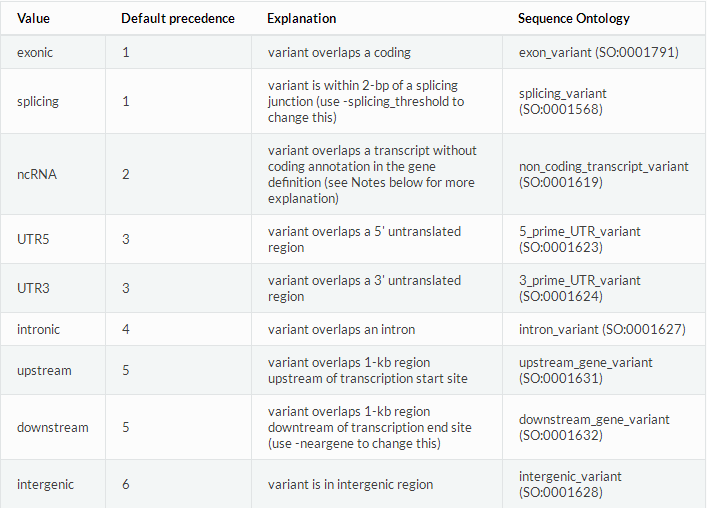
annovar.png 第二列信息则给突变位点所在的基因名称(如果突变在exonic/intronic/ncRNA),或者给出临近基因的名称
XX.exonic_variant_function给的信息略微多一点,包含了外显子的一些突变所导致的氨基酸的变化

第一列是变异所在的行号(input file文件);第二列是这些变异的functional consequences,包括有:nonsynonymous SNV, synonymous SNV, frameshift insertion, frameshift deletion, nonframeshift insertion, nonframeshift deletion, frameshift block substitution, nonframshift block substitution;其实这些annotation也有像上述优先级一样的表格,这里暂时没列出来,可参考官网文档
我顺便也查看了下我的
KPGP-00001_filtered_pass.snp.exonic_variant_function文件cut -f 2 KPGP-00001_filtered_pass.snp.exonic_variant_function |sort |uniq -c结果如下,并没有上述所说的很多注释信息,可能是由于只是Gene-based annotation吧:
32000 nonsynonymous SNV 1492 stopgain 80 stoploss 24804 synonymous SNV 245 unknown第三列则是基因名称、转录本ID以及其序列发生的变化
所以从上述可看出,如果单纯的用annovar的Gene-based annotation,其信息量其实并不多,只告诉了你突变所在或者临近的基因,以及一些简略的信息,之后需要好好用一下annovar更全面的功能
snpEFF
snpEFF的结果是在输入的vcf文件中添加了一个tag:ANN,我将这部分tag提取来,整理下,如下:
1 63268 rs75478250 T C ANN=C|non_coding_transcript_exon_variant|MODIFIER|OR4G11P|ENSG00000240361|transcript|ENST00000492842|unprocessed_pseudogene|1/1|n.321T>C||||||其信息相比annovar来说比较齐全(当然也跟我只用annovar的Gene-based annotation有关,不能怪软件。。),从non_coding_transcript_exon_variant可看出,snpEFF给出了每个变异位点的突变影响,用Sequence Ontology (SO)来表示;从上面的annovar的结果表格也可看出,其最后一列也列出了那些突变位置(exonic、splicing、ncRNA等)所对应的Sequence Ontology
其他所列的信息的含义以在之前博文中所记录了。。。
从snpEFF的snpEff_genes.txt和snpEff_summary两个结果文件可看出,我的测试文件中的effects type有20多种,那么应该怎么看这些Sequence Ontology (SO)呢,比如non_coding_transcript_exon_variant是什么意思
那么推荐一个网站:http://sequenceontology.org/browser/current_svn/term/SO:0001792,其实整篇博文最有用的就属这个网站了,其对non_coding_transcript_exon_variant这个含义做了详细的解释和说明,并标识其在不同变异注释软件中相对应的名称,最后还给了其Ontology父子节点是哪个
比如protein_protein_contact这个Sequence Ontology,单看字面意思,不能准确了解这个注释信息的含义,那么可以在上述网站查一下http://www.sequenceontology.org/browser/current_svn/term/SO:0001093
还有一个网站也蛮有用的,最初是从生信菜鸟团博客吐血推荐snpedia数据库,非常丰富的snp信息记录中看到的,可以查看每个dbSNP数据库中每个位点的详细信息;比如你用annovar或者snpEFF软件将vcf文件注释到dbSNP数据库后,发现一个比较重要的snp位点,但是想了解这些snp更为详细的信息,比如其纯合或者杂合突变有哪些影响、哪些文献报道了这些位点、这些位点在各个数据库中的ID是什么等等信息,那么就可以使用这个网站https://www.snpedia.com/index.php/SNPedia
比如rs671这个影响酒精代谢的突变位点,那么只需要进入网站https://www.snpedia.com/index.php/Rs671即可查看其详细信息,内容真的很丰富
本文出自于http://www.bioinfo-scrounger.com转载请注明出处