火山图(Volcano Plot)常用于展现差异表达的基因,横坐标常为Fold change(倍数),纵坐标为P value(P值),这里以静态和动态两种形势展示火山图,主要还是为了介绍下ggplot2的插件ggrepel和plotly包 #### 静态散点图(火山图)
比如现在有一个差异基因表达分析后的数据sample.txt,读入R
data <- read.table(file = "sample.txt", header = T, sep = "\t")
head(data)
##Row.names baseMean log2FoldChange lfcSE stat pvalue padj
##1 Cbl 1843.3501 1.720458 0.1308424 13.149091 1.72e-39 2.01e-35
##2 Nr3c1 130.6798 2.785015 0.2683346 10.378889 3.09e-25 1.80e-21
##3 Usp2 262.6009 -1.551694 0.1569736 -9.885063 4.83e-23 1.88e-19
##4 Reps2 1389.6760 1.506295 0.1696388 8.879425 6.72e-19 1.96e-15
##5 Atxn1 3045.2814 -1.221654 0.1406477 -8.685916 3.76e-18 8.77e-15
##6 Birc6 526.7531 -1.113232 0.1297528 -8.579636 9.52e-18 1.59e-14根据差异标准对每个基因进行分类UP(上调),DOWN(下调)和NOT(没有差异)
data$change <- as.factor(ifelse(data$padj < 0.01 & abs(data$log2FoldChange) > 1,ifelse(data$log2FoldChange > 1,'UP','DOWN'),'NOT'))然后接下来就是传统的ggplot2作图了
library(ggplot2)
p <- ggplot(data = data, aes(x = log2FoldChange, y = -log10(padj), color = change)) +
geom_point(alpha=0.8, size = 1) +
theme_bw(base_size = 15) +
theme(
panel.grid.minor = element_blank(),
panel.grid.major = element_blank(),
scale_color_manual(name = "", values = c("red", "green", "black"), limits = c("UP", "DOWN", "NOT"))
p
上述的常规的作图方法,当然如果想美观点可以在theme主题上进行设置,这里不展开了。如果我想了解某个点的gene symbol是什么,最先想到的是用ggplot2里面的geom_text函数将gene symbol在点周围标注上;当然不可能将所有点都标注,只标注差异表达基因的点
再给data数据库加一列sign信息,满足显著差异的赋值为基因ID,不显著差异的赋值为NA(这里的差异标准我随便设的)
data$sign <- ifelse(data$padj < 0.01 & abs(data$log2FoldChange) > 1,data$Row.names,NA)然后在上述图的代码基础上增加geom_text函数
p <- ggplot(data = data, aes(x = log2FoldChange, y = -log10(padj), color = change)) +
geom_point(alpha=0.8, size = 1) +
theme_bw(base_size = 15) +
theme(
panel.grid.minor = element_blank(),
panel.grid.major = element_blank(),
scale_color_manual(name = "", values = c("red", "green", "black"), limits = c("UP", "DOWN", "NOT")) +
geom_text(aes(label = sign), size = 3)
p
从上图可看出,由于点过于密集,导致那些点的标注已经产生了重叠,那么这时就能用ggrepel包的geom_text_repel函数了,比如用该函数替换上述代码的geom_text
library(ggrepel)
p <- ggplot(data = data, aes(x = log2FoldChange, y = -log10(padj), color = change)) +
geom_point(alpha=0.8, size = 1) +
theme_bw(base_size = 15) +
theme(
panel.grid.minor = element_blank(),
panel.grid.major = element_blank(),
scale_color_manual(name = "", values = c("red", "green", "black"), limits = c("UP", "DOWN", "NOT")) +
geom_text_repel(aes(label = sign), box.padding = unit(0.3, "lines"), point.padding = unit(0.4, "lines"), show.legend = F, size = 3)
p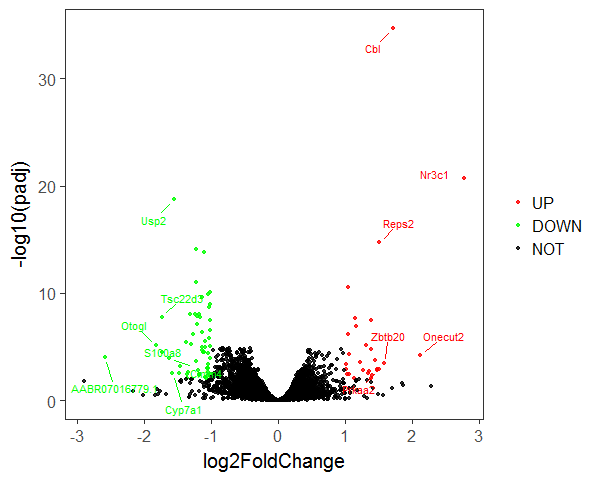
从上图可看出,一些密集的区域用线使标注各自原理,解决标注重叠的问题,蛮好用的
动态交互式(火山图)
上面讲的都是写静态的火山图,那么如果想与图有一定的交互,那么可以使用R的plotly包,以我初步对plotly包的理解,其可以分为两部分,一部分是以其自有的plot_ly函数画散点图,另一部分是通过该包对ggplot2的借口,用ggplotly函数将ggplot2作图结果转化为交互式
比如还是上面的数据(接上面的代码),先以plot_ly作图
library(plotly)
p <- plot_ly(data,
x = ~log2FoldChange,
y = ~-log10(padj),
text = ~sign,
type = 'scatter',
mode = 'markers'
)
p
从上图可看出,只要鼠标点到某个点上,即可查看其信息,比如可以有X轴、Y轴以及标注等信息,具体交互图可点击链接查看)http://www.bioinfo-scrounger.com/data/volcano_plot1.html(因为我还没搞定如何将html形式的动态图,放到wordpress搭建的博客的文章页面内。。。
上述plotly包自带的plot_ly函数毕竟功能太是太全,还不能画出像静态图那般的火山图,因此可以使用ggplotly函数来转化ggplot2的结果
p <- ggplot(data = data, aes(x = log2FoldChange, y = -log10(padj), color = change)) +
geom_point(aes(text = sign), alpha=0.8, size = 1) +
theme_bw(base_size = 15) +
theme(
panel.grid.minor = element_blank(),
panel.grid.major = element_blank(),
plot.title = element_text(hjust = 0.5)) +
scale_color_manual(name = "", values = c("red", "green", "black"), limits = c("UP", "DOWN", "NOT"))
p <- ggplotly(p)
p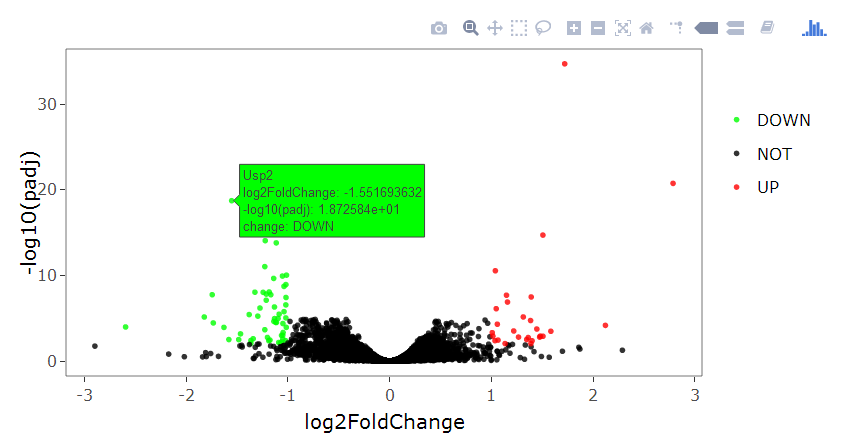
从上图结果可看出,ggplotly函数是不是很赞!框内分别显示了gene symbol、foldchange、p value以及上下调,想看哪个就点哪个,如果想看看交互式结果,可点这个链接查看http://www.bioinfo-scrounger.com/data/volcano_plot2.html
参考资料: https://zhuanlan.zhihu.com/EasyCharts-R
本文出自于http://www.bioinfo-scrounger.com转载请注明出处